標準graphicsだと単純なヒストグラム以外は、実用上、問題が出てくるので、ggplot2などのパッケージの利用を推奨します。
1 ヒストグラム
まずはirisデータセットの萼片の長さを使って、棒グラフの次に習うヒストグラムをプロットしましょう。
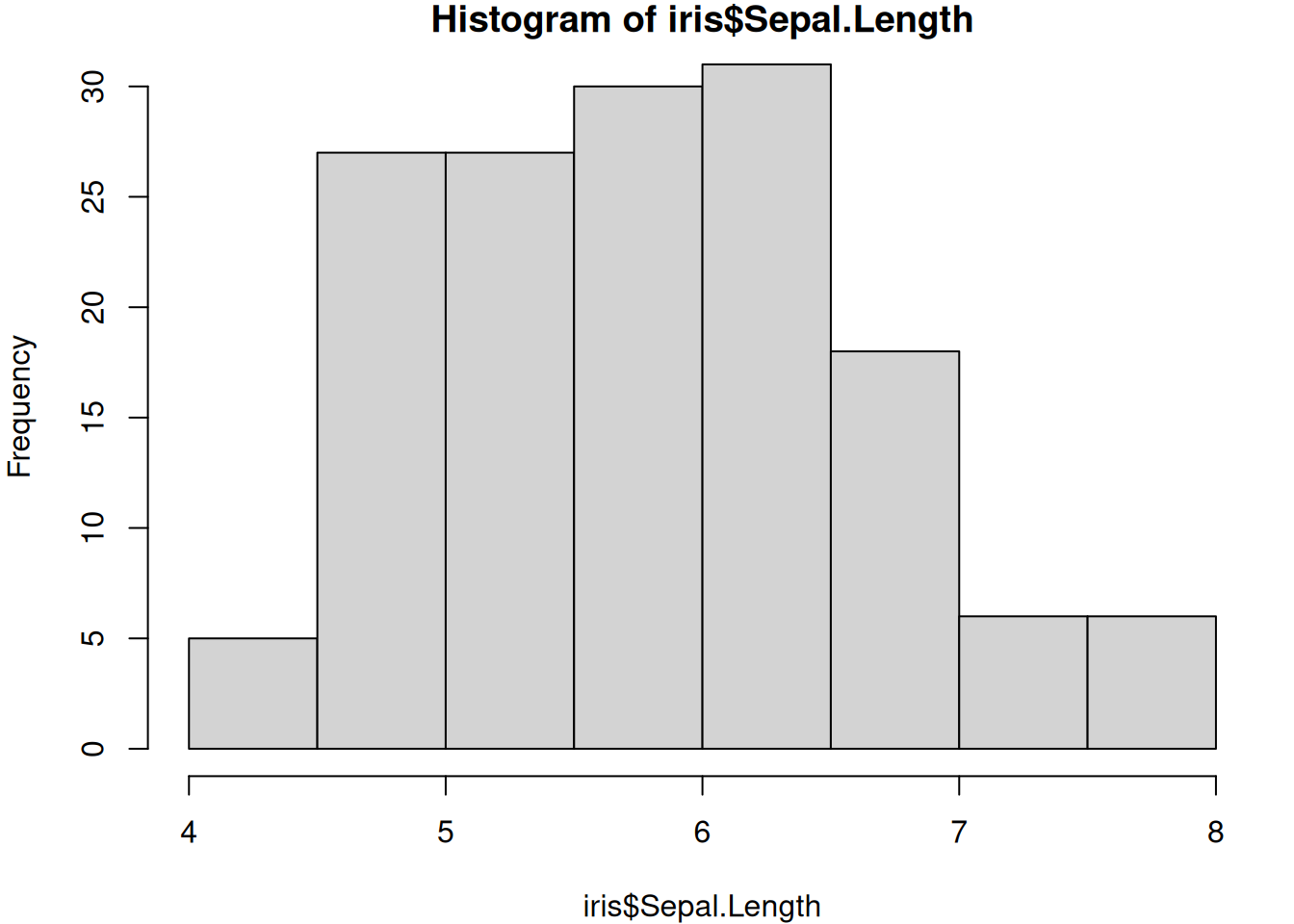
はい、できました。ドキュメントに入れずとも、確認でよく使うので便利です。
配色や軸の表示はplotなどと同様の引数で変えられます。
1.1 間隔(bins)
hist関数には癖が2つあります。ひとつは、引数dataがとれません。データフレームの列を複数使うときは、with演算子を使いましょう。もうひとつは、間隔(bins)の調整です。
省略時は自動設定してくれて、breaks引数でbinsを指定できますが、自動調整されるために思ったとおりにいかないので、閾値を指定した方が無難です。ただし、データの最小値と最大値が覆えるようにしないとエラーになるので注意してください。
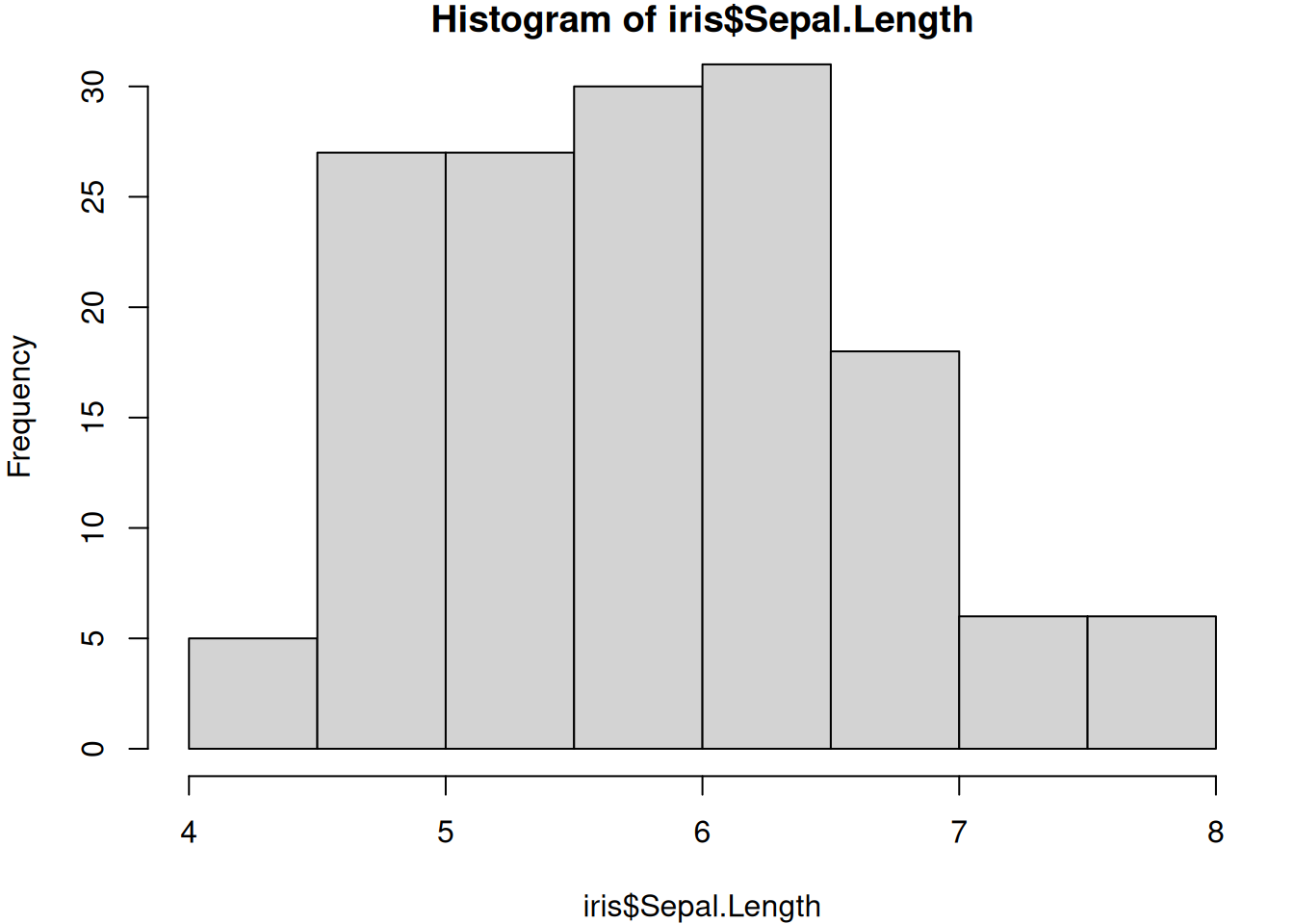
$breaks
[1] 4.0 4.5 5.0 5.5 6.0 6.5 7.0 7.5 8.0
$counts
[1] 5 27 27 30 31 18 6 6
$density
[1] 0.06666667 0.36000000 0.36000000 0.40000000 0.41333333 0.24000000 0.08000000
[8] 0.08000000
$mids
[1] 4.25 4.75 5.25 5.75 6.25 6.75 7.25 7.75
$xname
[1] "iris$Sepal.Length"
$equidist
[1] TRUE
attr(,"class")
[1] "histogram"戻り値のオブジェクト(リスト)のメンバーから、頻度(counts)と相対頻度(density)が計算できます。引数にplot = FALSEをつけると、描画せずに戻り値を戻します。
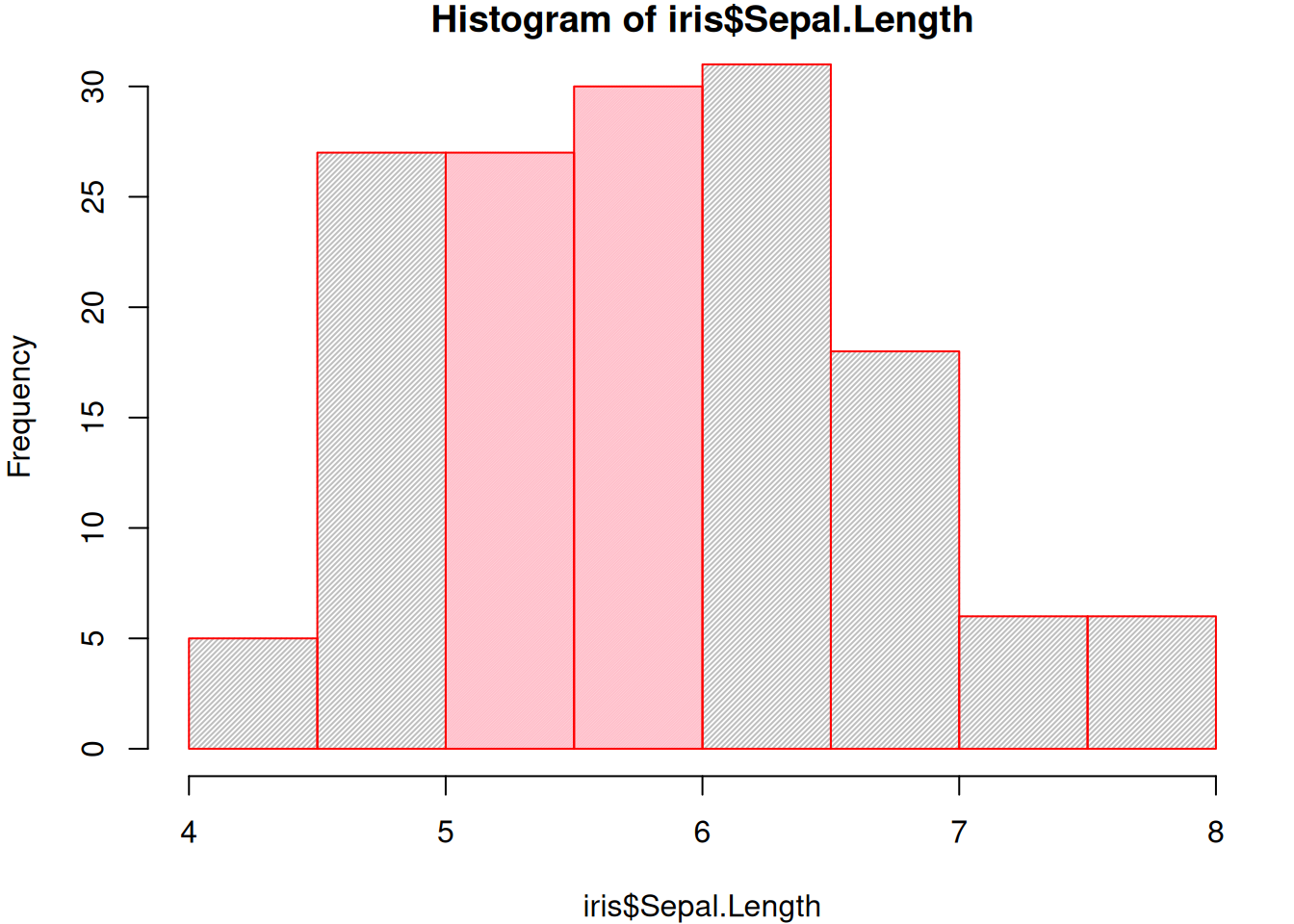
1.2 装飾
plotと同様に題名や軸の表示などが変えられる1他、barplotと同様に棒の塗りつぶしの色などを変更できます。
b <- seq(4, 8, 0.5)
col <- rep("gray", length(b))
col[5<=b & b<6] <- "pink"
d <- rep(50, length(b))
d[5<=b & b<6] <- 100
hist(iris$Sepal.Length, breaks = b,
border = "red",
col = col, # 塗りつぶし色
density = d, # 塗りつぶしの粗さ
angle = 45, # 塗りつぶしの角度
)
1.3 観測値
低水準関数rugを使うと、観測値を記入できます。後述する相対頻度のプロットで試します。
1.4 相対頻度
引数にfreq = FALSEをつけると、度数ではなく相対頻度をプロットします。見た目は軸の表示が変わるだけですが、予測値を追記しやすくなります。
with(iris, {
hist(Sepal.Length, breaks = seq(4, 8, 0.25), freq = FALSE, density = 50)
rug(Sepal.Length)
lines(density(Sepal.Length, bw = 0.25))
})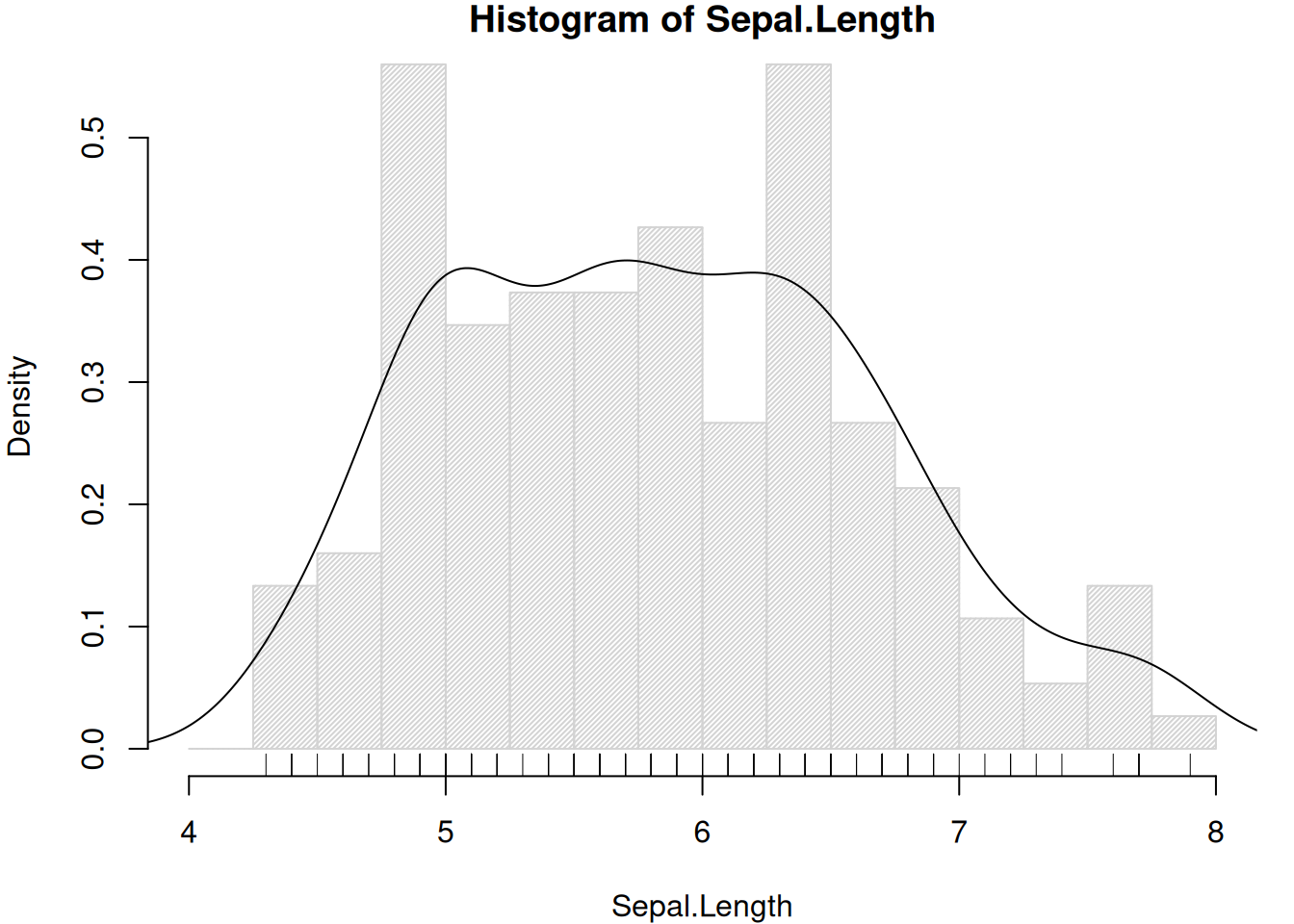
rugは標準ではヒストグラムがプロットされない0未満の領域に線で、観測値を一次元プロットする低水準関数です。
densityはカーネル密度推定をして、plotやlinesに渡して描画することができるオブジェクトを戻す関数です。標準では内部でkernel関数を呼び出しています。
1.5 複数ヒストグラムの同時プロット
工夫をすれば複数ヒストグラムの同時プロットもできます。irisデータセットをアヤメの種類ごとにヒストグラムを分けてみましょう。
with(iris, {
s <- split(Sepal.Length, Species)
b <- seq(4, 8, 0.25) # bins
d <- c(50, 25, 50) # 塗りつぶしの濃度
a <- c(45, 90, -45) # 塗りつぶしの角度
col <- c("gray", "blue", "pink")
for(i in 1:length(s)){
hist(s[[i]], breaks = b, col = col[i],
density = d[i], angle = a[i],
xlab = "Sepal.Length", main = "Iris", add = 1!=i)
}
legend("topright", names(s), fill = col, density = d, angle = a)
})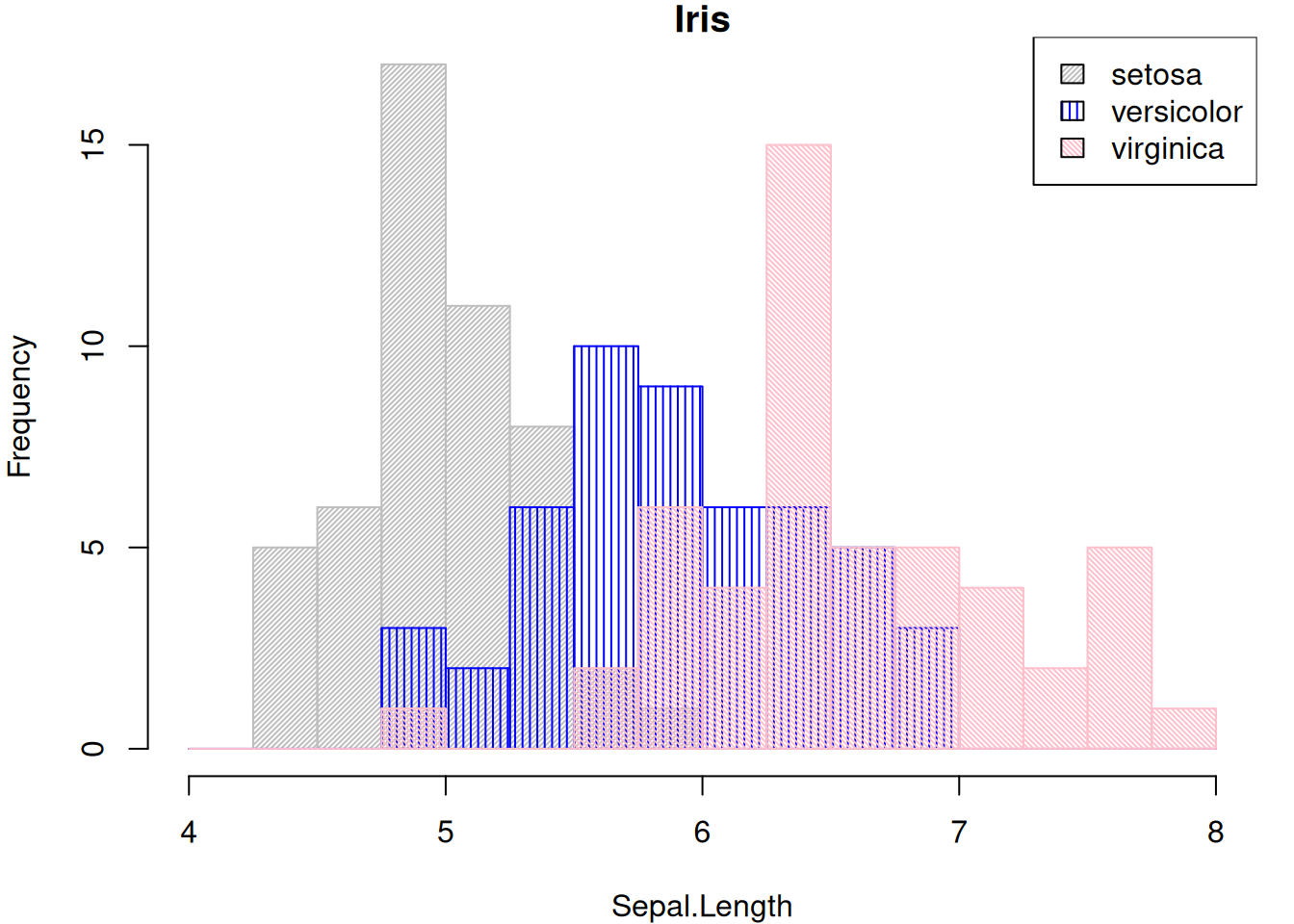
群間比較という意味では、boxplotを使ったほうが分かりやすいと思います。
2 カテゴリカルデータ
数値ではなく因子型の場合、ヒストグラムで頻度をプロットできません。1次元の場合は棒グラフ、2次元以上の場合はプロットではなく分割表を用いることが一般的ですが、カテゴリカルデータのためのプロットもあります。
2.1
ヒートマップ(heatmap)
2次元の行列で分割表が整理されていればヒートマップが使えます。髪と瞳の色のデータ・セットで試してみましょう。
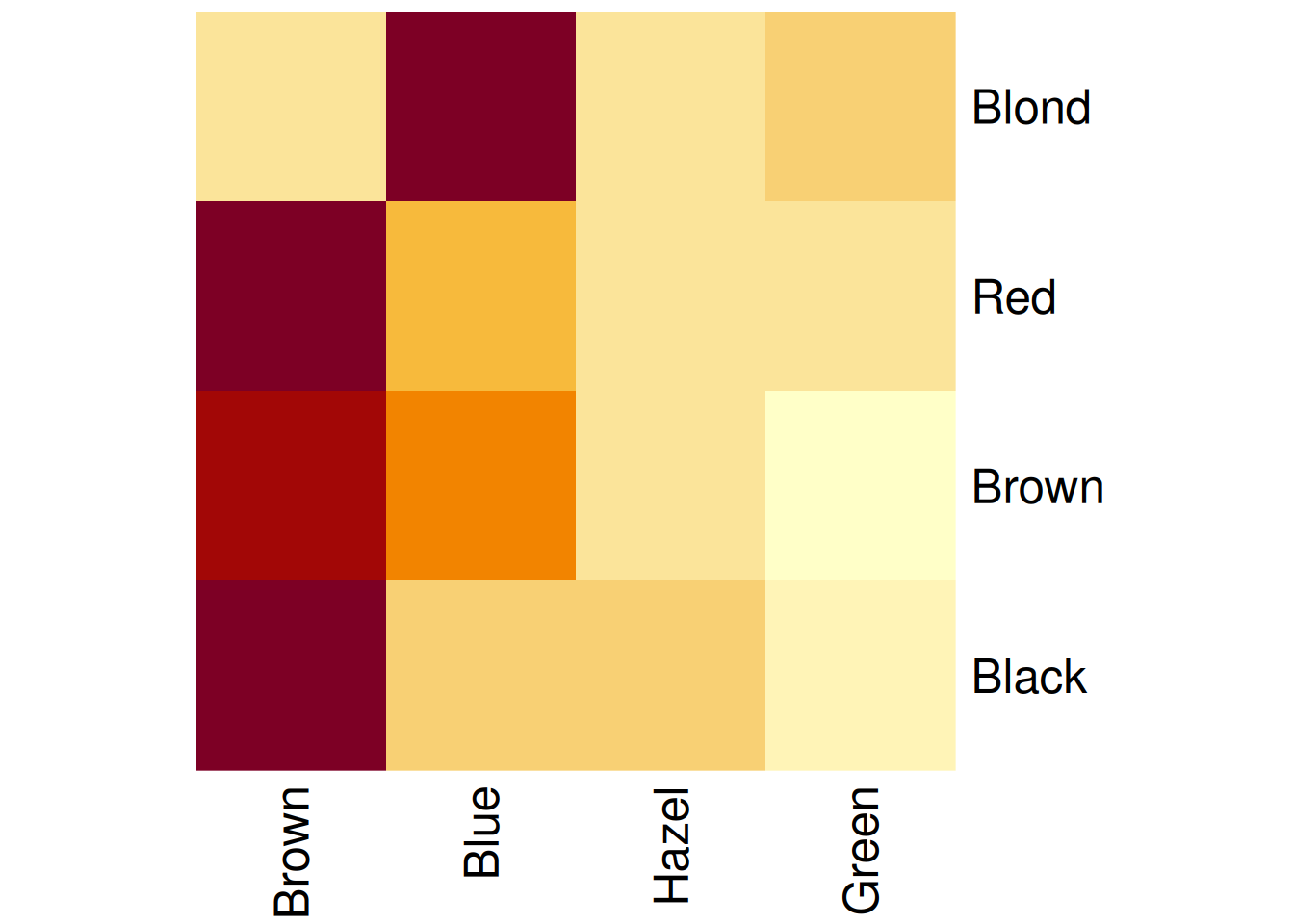
margin.tableは、分割表の次元を削減する関数です。データが3次元なので、削減しています。RowvとColvはクラスター分析に関するオプションで、NAを入れないと樹形図が表示されます。
2.1.1 見た目の変更
ある程度は見栄えを変更することができます。
X <- margin.table(HairEyeColor, 1:2)
labels <- names(dimnames(X))
crp <- colorRampPalette(c("white", "brown"))
heatmap(X, Rowv = NA, Colv = NA,
scale = "none", # 標準化なしで色分け
col = crp(max(X) + 1), # グラデーションに使う色
cexRow = 1.25, cexCol = 1.25, # フォントサイズ
margins = c(6, 6), # 余白サイズ
xlab = labels[2], ylab = labels[1])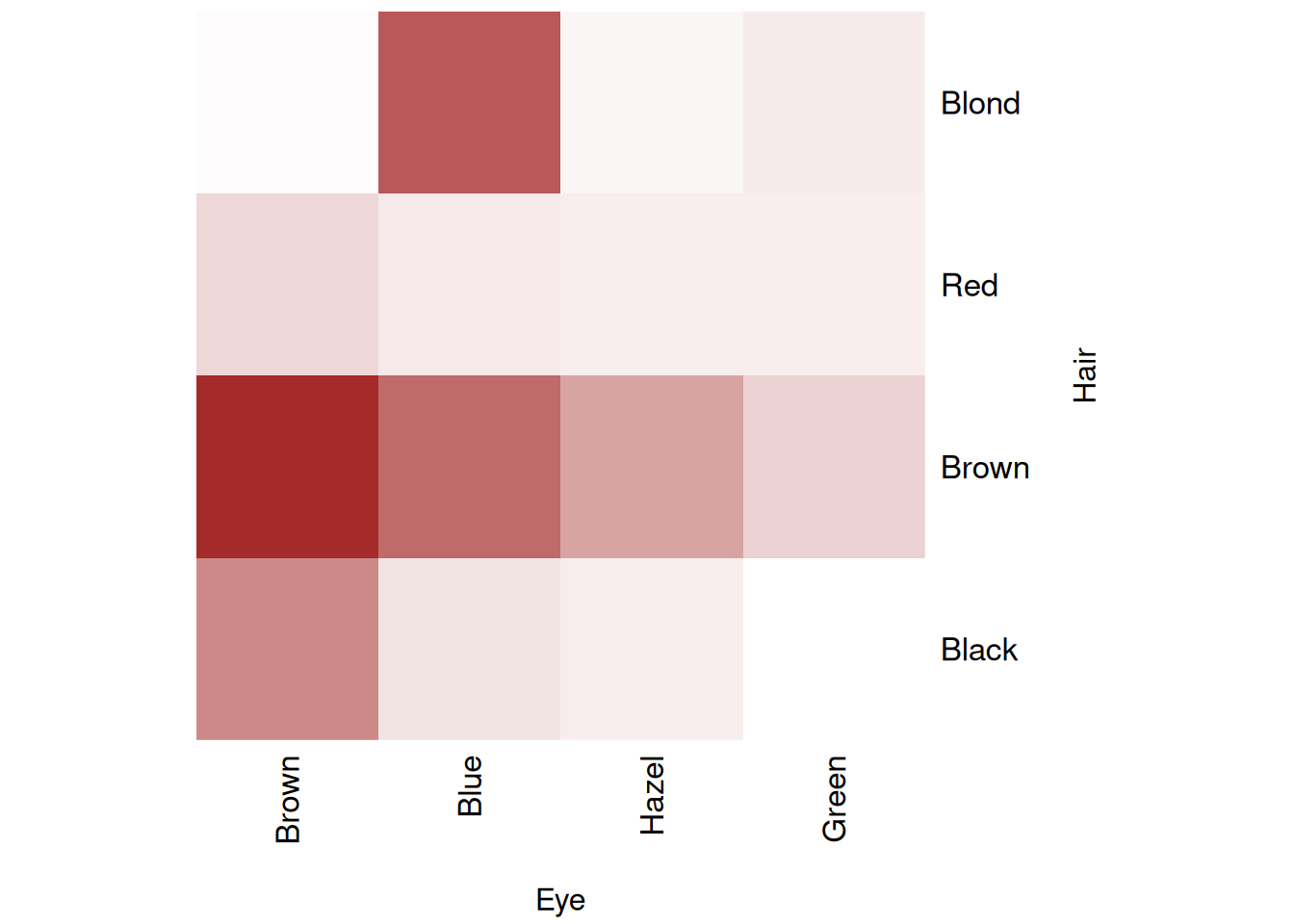
2.1.2 クラスター分析
デフォルトでは完全連結法で行と列をそれぞれクラスタリングをして樹形図も描きます。しかし難があって、表示は行ごとに数値を標準化しているのに、クラスタリングには反映されません。引数scaleは、色分けにしか作用しないのです。樹形図がヒートマップに対応しないことになります。
距離関数とクラスター化関数を指定できるので、手法の変更が可能です。列ごとに標準化したあと、群平均法で列をクラスタリングしてみましょう。なお、行が髪の色、列が瞳の色になっているので、列で標準化すると、瞳が青い人はブロンドが多いような解釈になります。
heatmap(X, Rowv = NA, Colv = NULL,
dist = \(X){
margin <- 2 # 1だと行ごと,2だと列ごと
m <- apply(X, margin, mean) # 列ごとの平均
s <- apply(X, margin, sd) # 列ごとの標準偏差
sX <- apply(X, c(2, 1)[margin], \(x) (x-m)/s) # 標準化
sX[is.na(sX)] <- 0 # 標準偏差0でNAのときは0に置換
if(2 == margin) sX = t(sX)
dist(sX)
}, hclustfun = \(d){
hclust(d, method = "average")
},
scale = "col", # 列で標準化してから色分け
col = crp(max(X) + 1),
cexRow = 1.25, cexCol = 1.25,
margins = c(6, 6),
xlab = labels[2], ylab = labels[1])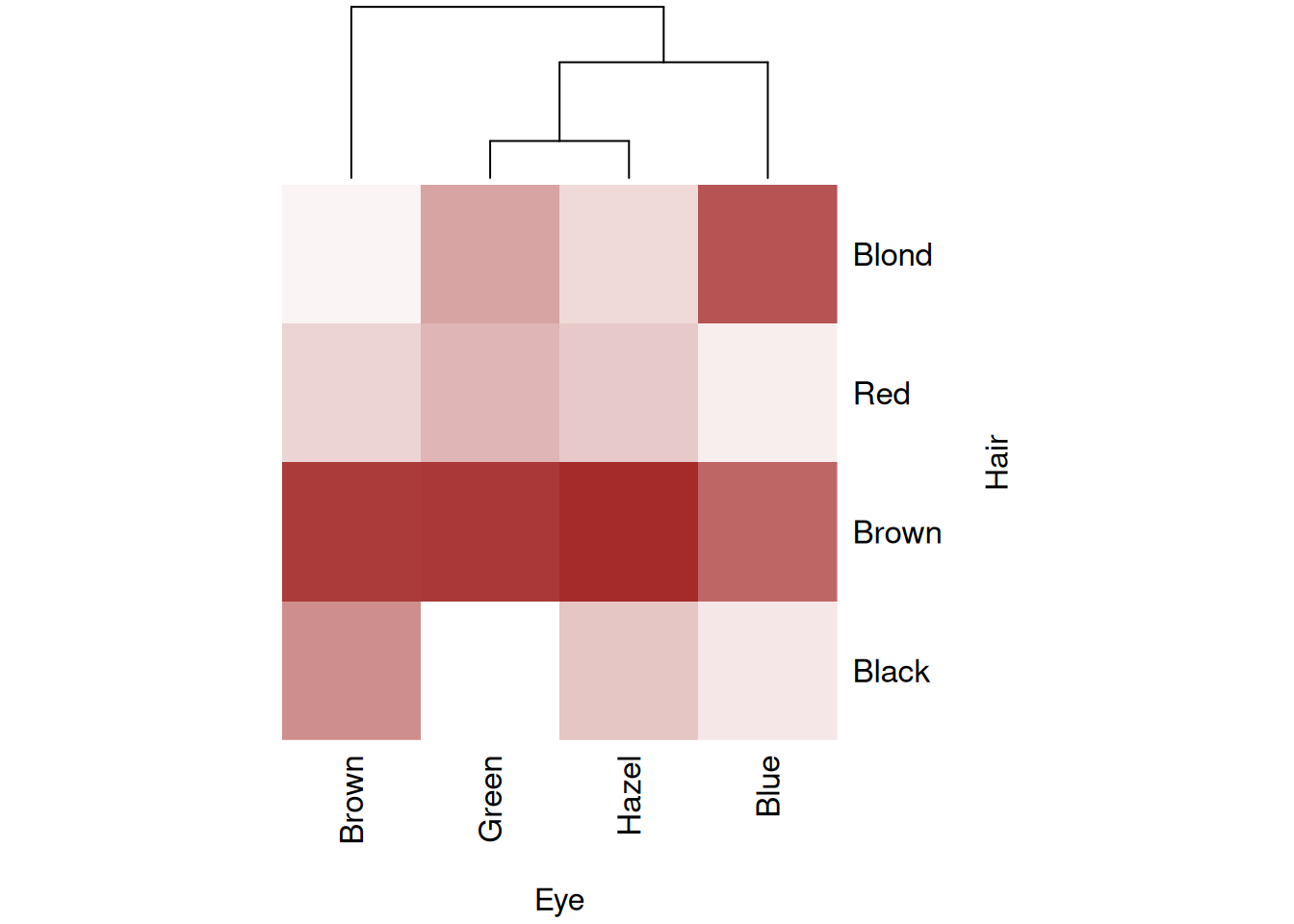
2.1.3 独立性検定
独立性検定をかける場合は、Rでチマチマとクロス集計と独立性検定を参照してください。
2.2
モザイクプロット(mosaicplot)
3次元データの場合は、mosaicplotで図示できます。集計データが配列になっている必要があるので、xtabsなどで集計した値を表示します。
集計済みのHairEyeColorを表示してみましょう。
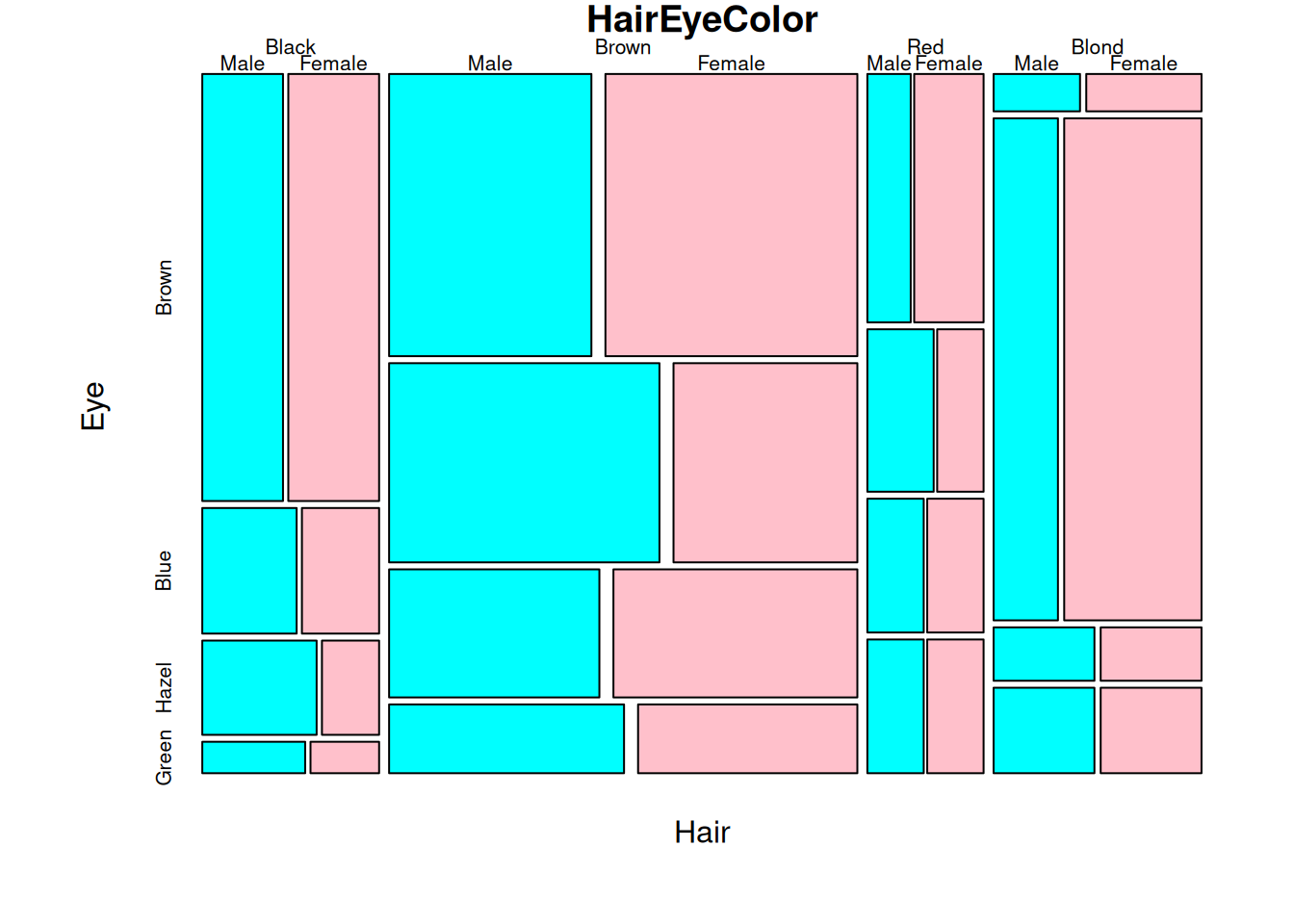
色分けは、dimnamesで見て最後の次元で行われます。
縦軸が瞳の色、横軸が髪の色、横軸のサブカテゴリーが性別となっています。
shade = TRUEをつけるかshade = c(1, 2, 3)のように要素が5つ以下の正の値のベクトルを与えると、loglin関数の予測値から計算された標準化残差で色分けします。計算方法は引数typeで変えられ、標準ではPearsonの\(\chi^2\)検定が用いられる残差が使われます。なお、colは無視され、色の指定はできないです。
2.3
連続変数の条件付きプロット(cdplot)
気温が低いとゴムが固くなって部品が破損するような、連続変数の条件付き二項分布に従うような場合は、cdplotを使います。ヘルプに載っている例そのものですが、
# スペースシャトルの部品O-ringの破損と気温の関係
fail <- factor(c(2, 2, 2, 2, 1, 1, 1, 1, 1, 1, 2, 1, 2, 1, 1, 1, 1, 2, 1, 1, 1, 1, 1),
levels = 1:2, labels = c("no", "yes"))
temperature <- c(53, 57, 58, 63, 66, 67, 67, 67, 68, 69, 70, 70, 70, 70, 72, 73, 75, 75, 76, 76, 78, 79, 81)
cdplot(fail ~ temperature)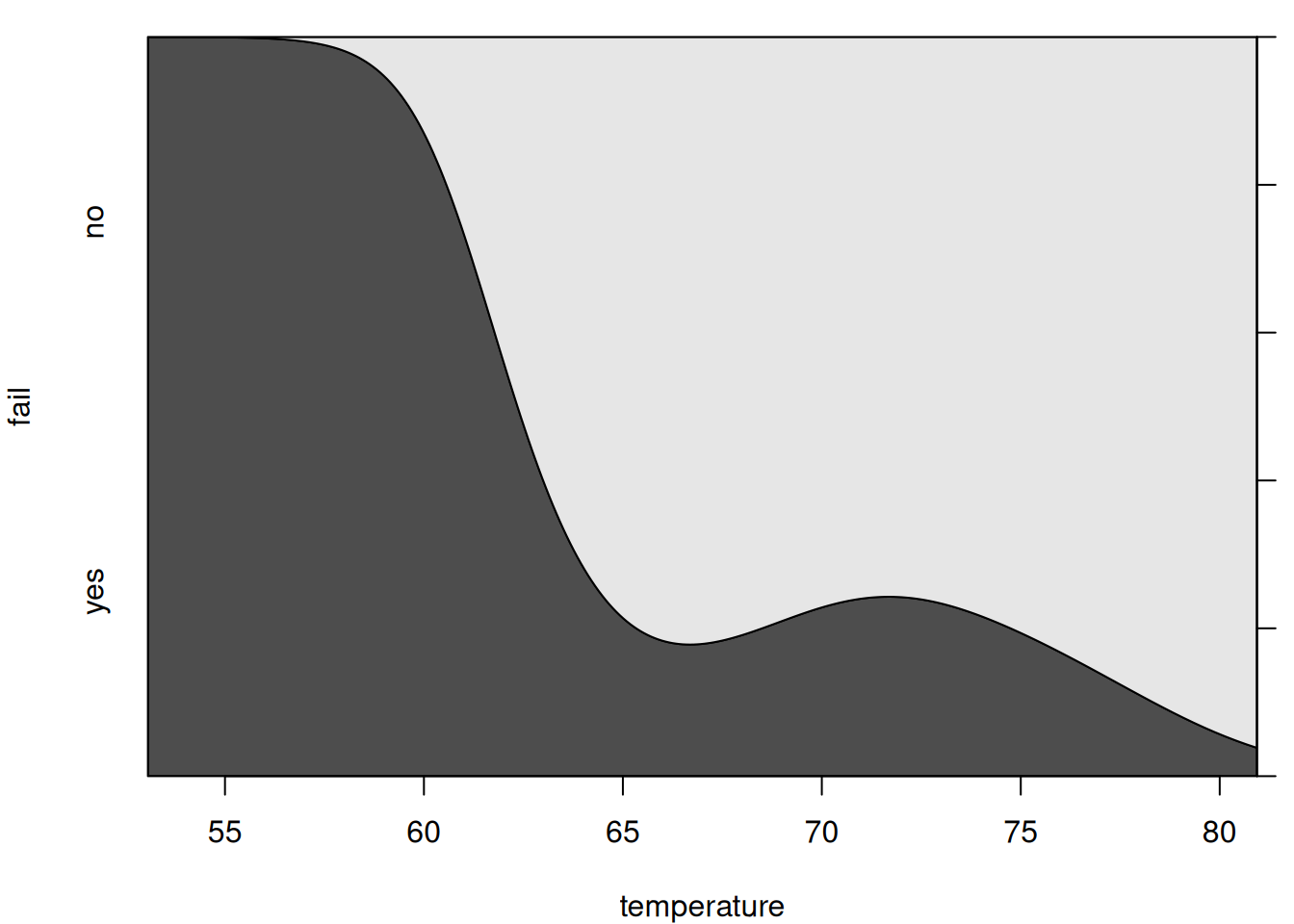
プロットすれば一目瞭然ですね。ポアソン分布や順序ロジットに従うような分布にも向いています。
従属変数が因子型2になることには注意してください。labelsを設定しておくと、描画で使われます。
滑らかになっているのは、内部的にカーネル密度推定を行っているからです。よりデータを忠実に描画したい場合は、spineplotでスパイログラムを描きます。横軸のスケールが非直観的なので、見せる相手は選びましょう。
Rのグラフィカル・オプションと低水準描画関数を参照してください。↩︎
因子型と順序型のざっとした説明の知識で間に合います。↩︎
