統計解析手法の説明や学習のためのデータセットとしては統計学の大家であるロナルド・フィッシャーの1936年の論文で使われた、もともとは植物学者のエドガー・アンダーソンが整備したアヤメ(Iris)のデータセットが長らく愛用されて来ました。最近は、他にも様々なデータセットが使われているのですが、パルマーペンギンのデータセットが人気のようです。これでRの集計関数を確認していきましょう。
2 パッケージのロード
palmerpenguins::penguinsとしてもアクセスできるのですが、ロードします。
標準のデータフレームではなくtibble形式で提供されていますが、今回の範囲では標準のデータフレームと同様に利用できます。
3 列名の確認
billがくちばしで、flipperがひれ、body massが体重になります。
[1] "species" "island" "bill_length_mm"
[4] "bill_depth_mm" "flipper_length_mm" "body_mass_g"
[7] "sex" "year" 4 因子の種類
因子データはlevelsでどんな種類があるのか確認できます。
[1] "Biscoe" "Dream" "Torgersen"[1] "Adelie" "Chinstrap" "Gentoo" [1] "female" "male" 5 集計
平均値をとってみましょう。
[1] 4201.754列(ベクトル)とna.rmを指定します。na.rmは欠損値を無視するかどうかのオプションで、デフォルトではFALSEで欠損値があるとエラーが出ます。
標準偏差(sd)、分散(var)、合計(sum)、最小値(min)、最大値(max)、中央値(median)などは同様に扱えます。尖度と歪度を確認したい場合は、momentsパッケージのkurtosisとskewnessを使うのが楽です。
rangeは最小値と最大値を同時に求めることができます。
[1] 2700 6300四分位をもとめるquantileは、実は任意の点を求める事ができます。
0% 25% 50% 75% 100%
2700 3550 4050 4750 6300 0% 5% 95% 100%
2700 3150 5650 6300 データが無限にあるわけではないので、quantileの計算結果は概算です。具体的な計算方法は?quantileで詳しく読めます。
6 クロス集計
何次元でも同様ですが、一次元と二次元の場合を試します。
6.1 一次元
因子ごとの頻度を調べる場合は、xtabs1が便利です。
island
Biscoe Dream Torgersen
168 124 52 species
Adelie Chinstrap Gentoo
152 68 124 sex
female male
165 168 頻度から比率を求めることもできます。
sex
female male
165 168 sex
female male
0.4954955 0.5045045 モデル式で変数を指定していますが、
species
Adelie Chinstrap Gentoo
558800 253850 624350 左辺を指定することで、その合計値を求めることもできます。
合計以外の平均などの場合は、aggregateを使うのが簡単です。
species body_mass_g
1 Adelie 3700.662
2 Chinstrap 3733.088
3 Gentoo 5076.016ベクトルの集計をとる関数であれば何でも用いることができ、ユーザー定義関数も入ります。
species body_mass_g
1 Adelie 1925
2 Chinstrap 2100
3 Gentoo 2350na.rmはデフォルトでTRUEが指定されます。
6.2 多次元
モデル式の右辺の変数を + で連結して増やせば、多次元の集計がとれます。
xtabsで因子ごとの頻度を計算してみましょう。
sex
species female male
Adelie 73 73
Chinstrap 34 34
Gentoo 58 61xtabsのクロス表の行や列ごとの合計は、addmargins関数で追加できます。
sex
species female male Sum
Adelie 73 73 146
Chinstrap 34 34 68
Gentoo 58 61 119
Sum 165 168 333合計がデフォルトですが、FUN = meanのように関数を指定することで、合計以外を付け加えることもできます。
aggregateで因子ごとの平均を求めるのも同様です。
species sex body_mass_g
1 Adelie female 3368.836
2 Chinstrap female 3527.206
3 Gentoo female 4679.741
4 Adelie male 4043.493
5 Chinstrap male 3938.971
6 Gentoo male 5484.836また、左辺の変数をcbindでつなげば、同時に複数の変数の集計が出せます。
# 集計する(mean関数を使っているので平均値が出る)
aggregate(cbind(flipper_length_mm, body_mass_g) ~ species + sex, penguins, mean) species sex flipper_length_mm body_mass_g
1 Adelie female 187.7945 3368.836
2 Chinstrap female 191.7353 3527.206
3 Gentoo female 212.7069 4679.741
4 Adelie male 192.4110 4043.493
5 Chinstrap male 199.9118 3938.971
6 Gentoo male 221.5410 5484.836なお、aggregateはdata.frameオブジェクトを戻すので、戻り値にaddmarginsは使えません。
6.3 プロット
なお、クロス集計もざっと見て異常がないか確認する場合などは、数字を見ていくよりも視覚化した方がわかりやすいです。
箱ひげ図を見ると異常値が入っているのとか、サブサンプル間の大きな違いがあれば把握できます。オプションですが箱の太さでサンプルサイズの違いもチェックできます。
boxplot(body_mass_g ~ sex + species, data = penguins, varwidth = TRUE, col = rep(c("pink", "skyblue"), 3))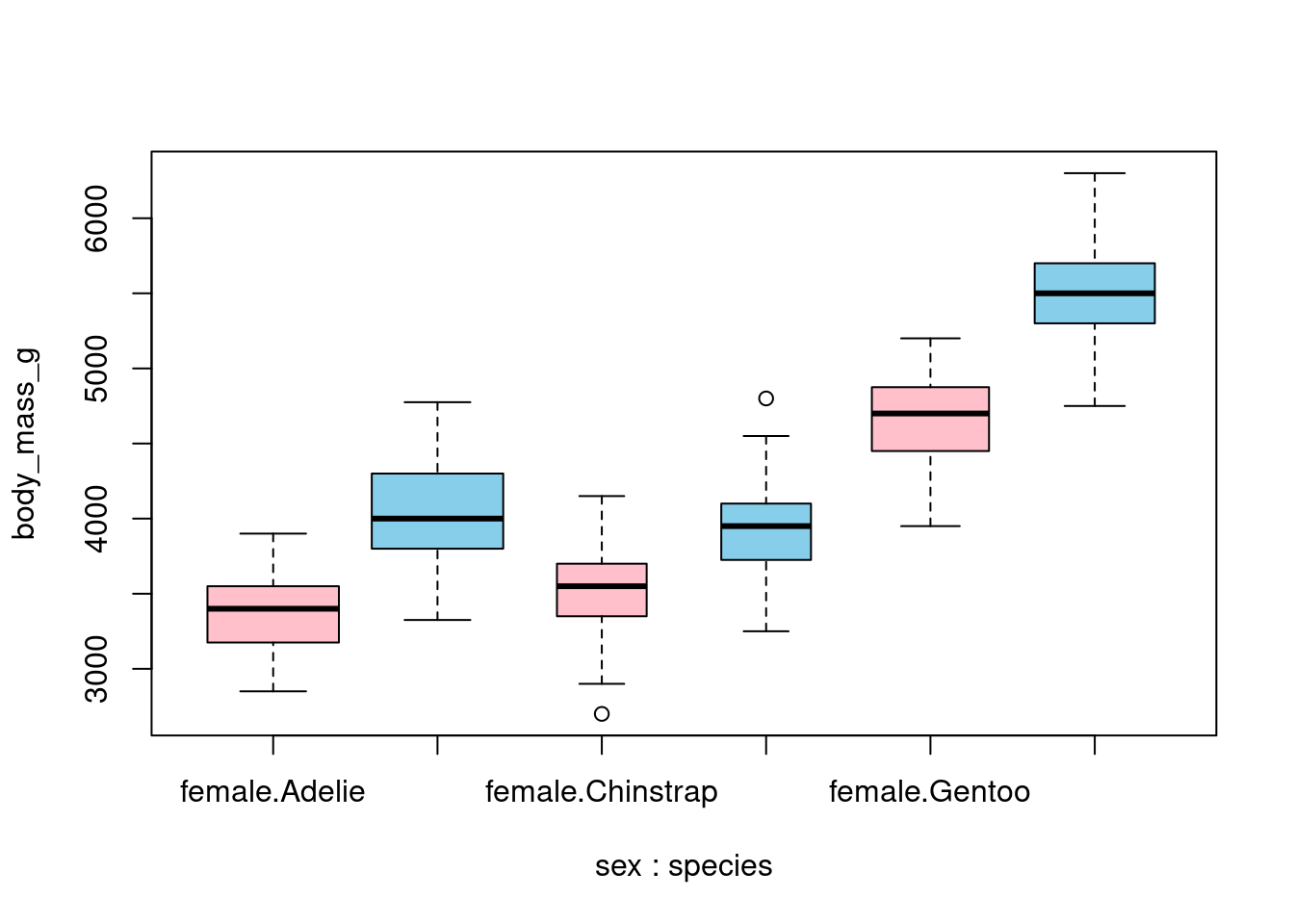
7 データフレームまるごとの要約統計量
実はここまで計算してきた統計量の多くは、データフレームにsummaryをかけるとまとめて計算してくれます。
species island bill_length_mm bill_depth_mm
Adelie :152 Biscoe :168 Min. :32.10 Min. :13.10
Chinstrap: 68 Dream :124 1st Qu.:39.23 1st Qu.:15.60
Gentoo :124 Torgersen: 52 Median :44.45 Median :17.30
Mean :43.92 Mean :17.15
3rd Qu.:48.50 3rd Qu.:18.70
Max. :59.60 Max. :21.50
NA's :2 NA's :2
flipper_length_mm body_mass_g sex year
Min. :172.0 Min. :2700 female:165 Min. :2007
1st Qu.:190.0 1st Qu.:3550 male :168 1st Qu.:2007
Median :197.0 Median :4050 NA's : 11 Median :2008
Mean :200.9 Mean :4202 Mean :2008
3rd Qu.:213.0 3rd Qu.:4750 3rd Qu.:2009
Max. :231.0 Max. :6300 Max. :2009
NA's :2 NA's :2 次の計算に再利用しない場合は、これが手っ取り早いです。
8 まとめ
Palmer Penguins datasetを用いてRの集計関数をざっと見て来ました。Iris datasetでは因子は品種の1つしかない一方、Palmer Penguins datasetは観察地点、種類、性別の3つの因子があるので、モデル式で複数の因子を簡単に指定できることを示せました。
base::tableでも計算できますが、こちらはモデル式とデータフレームの指定ができません。↩︎
